Pyruvate déshydrogénase
| Pyruvate déshydrogénase | |||||||||||||||||||
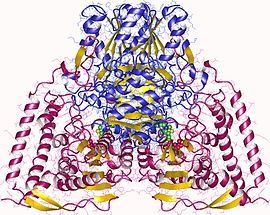 Modélisation d'un hétérotétramère de pyruvate déshydrogénase humaine avec les deux sous-unités α en violet, les deux ions Mg2+ en vert foncé, les deux TPP en orange-vert-rouge-bleu, les deux sous-unités β en bleu, et les deux ions K+ en magenta (PDB 1NI4[1]). | |||||||||||||||||||
| Caractéristiques générales | |||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Nom approuvé | Pyruvate déshydrogénase (lipoamide) | ||||||||||||||||||
| Symbole | PDH | ||||||||||||||||||
| N° EC | 1.2.4.1 | ||||||||||||||||||
| Gène PDHA1 – Sous-unité α1 | |||||||||||||||||||
| Homo sapiens | |||||||||||||||||||
| Locus | Xp22.12 | ||||||||||||||||||
| Masse moléculaire | 43 296 Da[2] | ||||||||||||||||||
| Nombre de résidus | 390 acides aminés[2] | ||||||||||||||||||
| Liens accessibles depuis GeneCards et HUGO. | |||||||||||||||||||
| |||||||||||||||||||
| Gène PDHA2 – Sous-unité α2 | |||||||||||||||||||
| Homo sapiens | |||||||||||||||||||
| Locus | 4q22.3 | ||||||||||||||||||
| Masse moléculaire | 42 933 Da[2] | ||||||||||||||||||
| Nombre de résidus | 388 acides aminés[2] | ||||||||||||||||||
| Liens accessibles depuis GeneCards et HUGO. | |||||||||||||||||||
| Gène PDHB – Sous-unité β | |||||||||||||||||||
| Homo sapiens | |||||||||||||||||||
| Locus | 3p14.3 | ||||||||||||||||||
| Masse moléculaire | 39 233 Da[2] | ||||||||||||||||||
| Nombre de résidus | 359 acides aminés[2] | ||||||||||||||||||
| Liens accessibles depuis GeneCards et HUGO. | |||||||||||||||||||
| |||||||||||||||||||
La pyruvate déshydrogénase (ou PDH) — à ne pas confondre avec la pyruvate décarboxylase[3] bien qu'on l'appelle parfois également ainsi — est la première des trois enzymes du complexe pyruvate déshydrogénase (PDC), constitué d'une décarboxylase, d'une acétyltransférase et d'une oxydoréductase intervenant séquentiellement pour catalyser la décarboxylation oxydative du pyruvate en acétyl-CoA, réaction qui assure notamment la liaison entre la glycolyse et le cycle de Krebs. Les autres enzymes de ce complexe sont la dihydrolipoamide S-acétyltransférase (E2) et la dihydrolipoyl déshydrogénase (E3).
La PDH est inhibée par la PDHK (ou pyruvate déshydrogénase kinase).
Le complexe pyruvate déshydrogénase
La réaction globale catalysée par le PDC est la suivante :

+ CoA-SH + NAD+ → NADH + H+ + CO2 + 
Pyruvate Acétyl-CoA
Le mécanisme de cette réaction est assez complexe, et peut être résumé par le schéma simplifié ci-dessous :

Mécanisme réactionnel du complexe pyruvate déshydrogénase (R = H sur ce schéma) :
- la pyruvate déshydrogénase (E1) catalyse les étapes A et B avec la thiamine pyrophosphate (TPP),
- la dihydrolipoamide S-acétyltransférase (E2) catalyse l'étape C avec le lipoamide et la coenzyme A (CoA-SH),
- la dihydrolipoyl déshydrogénase (E3) catalyse les étapes D et E avec la FAD et la NAD+.
La pyruvate déshydrogénase
| N° EC | EC 1.2.4.1 |
|---|---|
| N° CAS | 9014-20-4 |
| Cofacteur(s) | TPP |
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
| GO | AmiGO / EGO |
modifier 
La pyruvate déshydrogénase proprement dite (E1) catalyse deux réactions successives : la décarboxylation du pyruvate avec la thiamine pyrophosphate (TPP) comme cofacteur, et l'acétylation du lipoamide, ce dernier étant lié à la dihydrolipoamide S-acétyltransférase (E2).
 |  | |
| Thiamine pyrophosphate (TPP) | Lipoamide |
Le mécanisme de cette réaction peut être schématisé comme suit :

La forme de résonance ylure de la thiamine pyrophosphate (TPP) commence par attaquer la cétone électrophile du pyruvate. L'intermédiaire β-alcoolate se décarboxyle par la suite en un énol déprotoné sur l'atome de carbone pour former un dipôle 1,3 stabilisé avec un atome d'azote chargé positivement dans l'hétérocycle de la thiamine. Ce dipôle 1,3 est acétylé par le lipoamide lié à un résidu de lysine de la dihydrolipoamide S-acétyltransférase (E2).
La TPP est activée sur la pyruvate déshydrogénase par liaison covalente à un résidu glutamate (Glu-59 chez l'homme) en imposant une conformation en V dans laquelle l'atome N4' du cycle aminopyrimidine — le même cycle que celui de la cytosine — établit un pont hydrogène intramoléculaire avec l'atome C2 du groupe thiazole.
Notes et références
- ↑ (en) E. M. Ciszak et al. « Structural basis for flip-flop action of thiamin pyrophosphate-dependent enzymes revealed by human pyruvate dehydrogenase » dans J. Biol. Chem., 2002, 278, 21240-21246. DOI 10.1074/jbc.M300339200 PMID 12651851
- ↑ a b c d e et f Les valeurs de la masse et du nombre de résidus indiquées ici sont celles du précurseur protéique issu de la traduction du gène, avant modifications post-traductionnelles, et peuvent différer significativement des valeurs correspondantes pour la protéine fonctionnelle.
- ↑ La pyruvate décarboxylase (EC 4.1.1.1) convertit le pyruvate en acétaldéhyde en vue de produire de l'éthanol par fermentation alcoolique.
Articles connexes
- α-Cétoglutarate déshydrogénase, enzyme E1 du complexe α-cétoglutarate déshydrogénase
- 3-Méthyl-2-oxobutanoate déshydrogénase, enzyme E1 du complexe 3-méthyl-2-oxobutanoate déshydrogénase
 Portail de la biochimie
Portail de la biochimie












